Pourquoi choisir Simplot++ ?
Motivation :
La détection précise de la similarité de séquence et de la recombinaison homologue sont des éléments essentiels de nombreuses analyses évolutives
Resultats :
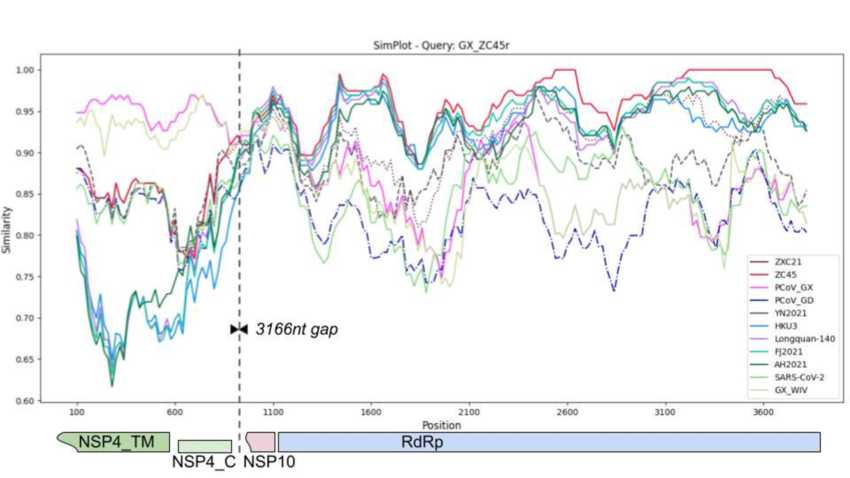
Nous avons développé SimPlot++, une application open-source multiplateforme implémentée en Python, qui peut être utilisée pour produire des graphiques de similarité de séquence de qualité de publication en utilisant 63 modèles de distance nucléotidique et 20 modèles de distance d'acides aminés, pour détecter des événements de recombinaison intergénique et intragénique en utilisant des tests Φ, Max-χ², NSS ou de proportion, et pour générer et analyser des réseaux interactifs de similarité de séquence. SimPlot++ prend en charge le traitement de données multicœur et fournit des diagnostics utiles de la calculabilité des distances.
Nous vous invitons à explorer notre site pour en savoir plus sur les fonctionnalités et l'utilisation de SimPlot++. Notre application offre une solution robuste et polyvalente pour les analyses évolutives, garantissant des résultats précis et fiables dans la détection de la similarité de séquence et de la recombinaison homologue. Grâce à son support multiplateforme et à son implémentation en Python, SimPlot++ est facilement accessible et utilisable sur différentes plateformes informatiques. Nous sommes convaincus que SimPlot++ sera un outil précieux dans vos travaux d'analyses évolutives. N'hésitez pas à le télécharger et à l'essayer par vous-même. Si vous avez des questions ou besoin d'assistance, notre équipe est toujours là pour vous aider. Nous vous remercions de votre intérêt pour SimPlot++. Nous avons hâte de voir comment notre application contribuera à vos recherches et analyses évolutives.